Procedimiento para la detección de recombinación homóloga in vivo analizando formas de gfp recombinantes
Sistema para la identificación, con microscopio de fluorescencia, de reordenaciones en el genoma (recombinación) originadas por diferentes condiciones. Emplea levaduras que expresan proteínas fluorescentes de diferente localización celular.
Estado de protección de la tecnología
Patente nº ES2610744.
¿Qué buscamos?
Se buscan empresas interesadas en la licencia de esta tecnología.
Facultad de Ciencias de la Salud. Centro de Investigaciones Científicas Avanzadas
Descripción
Nuestra tecnología (patente ES2610744) proporciona un sistema que permite identificar, con células vivas, el efecto tanto de agentes químicos como de mutaciones génicas con consecuencias en la reorganización de los genes y potencialmente cancerígenos. Esta identificación se realiza en el microscopio de fluorescencia con células sometidas a las condiciones que se quieran estudiar. El procedimiento permite obtener resultados en muy corto espacio de tiempo.
En la primera versión de éste análisis de recombinación se emplean dos genes modificados, en este caso al fusionarlos con proteína fluorescente verde (GFP), presentes en la levadura Saccharomyces cerevisiae. Dichos genes dan lugar a dos proteínas fluorescentes con diferente localización celular: una de ellas es nucleolar y emite una fluorescencia intensa; la otra emite una fluorescencia más tenue y es citosólica (véase la imagen 1). La recombinación entre los dos genes fusionados a GFP causa cambios en el patrón de fluorescencia celular (nucleolar versus citosólico) por la pérdida de uno de ellos (véase la imagen 2). Su frecuencia puede ser cuantificada.
Esta tecnología no implica grandes infraestructuras y se pueden analizar de un modo rápido las frecuencias de recombinación sin tener que hacer extracciones de ácidos nucleicos ni análisis moleculares. El procedimiento es aplicable a células vegetales y humanas. Estas últimas aplicaciones tienen cabida en medicina y biotecnología.
Otra información de interés:
Publicaciones y difusión relacionados con la tecnología:
- Varela-Rodríguez, B. M.; Alvarez-Felgar, T.; Rodríguez Torres, A. M. y Freire-Picos M. A. «A microscopy based system to detect homologous recombination in yeasts». Congreso BAC2017, León, julio de 2017, p. 105. ISBN: 978-84-947468-0-2.
- Varela Rodríguez, B. «Expresión de formas recombinantes de GFP en levaduras: efecto de la cepa y componentes del poro nuclear». Tesis de licenciatura, Facultad de Ciencias, UDC noviembre de 2013.
- Varela Rodríguez, B. «Expresión en levaduras de genes GFP recombinantes en el mutante Δnup84 del Complejo del Poro nuclear». Trabajo de fin de máster, Facultad de Ciencias, UDC, junio de 2014.
- Varela-Rodríguez, B. M.; Rodríguez Torres, A. M.; Álvarez, T. y Freire-Picos, M. A. «3’-Untranslated regions are involved in Nup84-dependent recombinant events». Artículo en prensa, 2018.
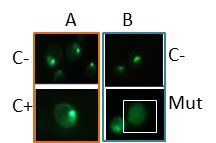
Representación esquemática de las posibles localizaciones celulares.
Ejemplo. A Célula no sometida a cambios (C- control negativo) (C+ control positivo con fluorescencia nucleolar y citosólica). B Experimento en células mutadas.
Valores añadidos
En el estado actual de desarrollo, esta tecnología permite identificar cambios originados por mutaciones celulares, agentes químicos externos o fármacos que causen reorganizaciones génicas, empleando levaduras como organismo modelo y sin necesidad de hacer ningún tipo de procesado celular. Únicamente mediante visualización por microscopía de fluorescencia.
Una gran ventaja frente a los estudios que aplican técnicas de PCR es que aquí las células se visualizan directamente tras cultivarlas, mientras que el análisis por PCR requiere el procesado de las muestras, la propia PCR y la electroforesis. Por otro lado, con nuestra técnica podemos identificar casos de recombinación en células individuales que podrían pasar desapercibidos, si su frecuencia es baja, en una PCR con el DNA de varias células.
Además, en el caso del estudio de recombinación por técnicas de genética clásica, es necesario detectar crecimiento celular en placas selectivas, lo que supone un gasto de tiempo (normalmente 2-3 días) y materiales de cultivo.
La tecnología es aplicable y está patentada para su uso en cualquier tipo de célula eucariota. Por tanto, en estos casos tampoco es necesario hacer extracciones de ADN ni PCR. Asimismo, permite diferenciar el sentido de la recombinación según se pierda la fluorescencia citosólica o la nucleolar. Incluye dos cepas de levadura para tener un control positivo y otro negativo en los estudios a los que se aplique.
Por último, esta tecnología permite el desarrollo de un software específico de análisis de imagen.
Aplicaciones por sector
Ejemplos de aplicación por sector:
- Investigación básica y/o aplicada: analizar in vivo el efecto causado por la mutación de un gen que afecte a la reorganización genómica. Esta aplicación ya ha sido validada por nuestro grupo de investigación y sirve para otras especies, plantas, animales y humanos.
- Medioambiental: estudiar el aumento de frecuencia de la recombinación celular causada por agentes potencialmente dañinos presentes en medios acuáticos, con las subsiguientes consecuencias negativas para los seres vivos.
- Farmacológico y biomédico: efecto de un medicamento o de un potencial fármaco, en la tasa de recombinación, que esté asociado con un aumento de las probabilidades de cáncer.
Grupo de investigación
-
- Expresión Génica en Levaduras
- (Levagen)
-
- Grupo Fisiopatología Endocrina, Nutricional y Médica
- (FENM)
Responsable
- María Angeles Freire Picos
- Ana Mª Rodríguez Torres
- Bárbara Mª Varela Rodríguez
Contacta con nosotros
-
-
Oficina de Transferencia de Resultados de Investigación
Edificio de Servicios Centrales de Investigación
Campus de Elviña, s/n
15071 A Coruña
-
Oficina de Transferencia de Resultados de Investigación
Última actualización
2019-07-16